DNA Replikation
Was ist die DNA Replikation, wie läuft sie bei Eukaryoten und Prokaryoten ab und welche Enzyme sind daran beteiligt? Das lernst du hier.
Du willst den Inhalt dieses Beitrags noch schneller verstehen? Kein Problem, dann schaue dir gerne unser anschauliches Video dazu an!
Inhaltsübersicht
DNA Replikation einfach erklärt
Wenn sich unsere Zellen aufgrund von Wachstum oder Fortpflanzung teilen (=Cytokinese ), muss sich auch der Zellkern teilen (Mitose ). Die dort enthaltenen genetischen Informationen müssen nämlich in Form von DNA an andere Zellen weitergeben werden.
Bevor die Kernteilung stattfindet, muss zunächst eine identische Kopie der DNA angefertigt werden. Diesen Vorgang kannst du auch als DNA Verdopplung oder DNA Replikation bezeichnen.
Hierfür sind zahlreiche Enzyme (u.a. DNA-Polymerase ) nötig. Die DNA, die normalerweise als Doppelhelix vorkommt, muss entwunden und die jeweiligen Wasserstoffbrückenbindungen zwischen den einzelnen DNA Basenpaaren müssen aufgetrennt werden. Das kannst du dir wie bei der Öffnung eines Reißverschlusses vorstellen. Die beiden offen gelegenen Einzelstränge stellen eine Vorlage (Matrize) für jeweils einen neu herzustellenden DNA Strang dar. An jede aufgetrennte Base kann sich jeweils ein DNA-Baustein (Nukleotid ) mit der entsprechenden passenden (komplementären) Base anlagern.
Dadurch entstehen zwei identische DNA-Doppelstränge, bei denen jeweils eine Hälfte von der ursprünglichen DNA stammt und eine Hälfte neu herstellt wurde. Deshalb kannst du auch von einer semikonservativen (lat. semi „halb“ und conservare „erhalten“) Replikation sprechen.
Die DNA Replikation (Reduplikation) (engl. dna replication) ist die identische Verdopplung des Erbguts (DNA). Sie wird in 3 Phasen (Initiation, Elongation und Termination) unterteilt und beginnt an einem definierten Startpunkt (Origin, Replikationsursprung).
Du willst dir einen Überblick über den Ablauf der DNA-Replikation verschaffen? Dann ist unser kurzer Beitrag zu den Grundlagen der Replikation, genau das Richtige für dich!
Semikonservative Replikation Prinzip
Unter Forschern herrschte Einigkeit, dass sich die DNA für die Weitergabe der genetischen Informationen verdoppeln muss. Doch nach welchem Prinzip diese identische Replikation verlaufen sollte, stand offen. Denkbar waren drei verschiedene Möglichkeiten: dispersiv, total konservativ oder halb konservativ.
Bei der konservativen Verdopplung würde ein DNA-Doppelstrang komplett erhalten bleiben und zusätzlich ein völlig neuer entstehen. Unter der dispersiven Verdopplung kannst du verstehen, dass sich alte und neue DNA Stücke entlang aller 4 Stränge verteilen.
Im sogenannten Meselson Stahl Experiment konnte dann aber eindeutig belegt werden, dass es sich bei der DNA Verdopplung um eine semikonservative Replikation handelt. Aus einer ursprünglichen DNA entstehen also zwei Moleküle, die sich jeweils zur Hälfte aus einem komplett „alten“ und einem komplett „neuen“ Strang zusammensetzen.
Ein paar Ausnahmen gibt es aber trotzdem: Die identische Replikation in der mitochondrialen DNA läuft nach einem anderen Mechanismus, dem sogenannten D-Loop Prozess (engl. displacement loop: Ablöse- oder Verdrängungsschlaufe), ab. Ebenso wie die Verdopplung der Erbinformation in Viren und Plasmiden , bei denen ringförmige DNA-Einzelstränge (ssDNA) vorliegen. Hier findet ein völlig anderer Mechanismus statt: Die Rolling Circle Replication („Rollender-Ring Replikation“).
Studyflix vernetzt: Hier ein Video aus einem anderen Bereich
DNA Replikation Ablauf im Überblick
Schauen wir uns nun aber den Ablauf der semikonservativen DNA Replikation im Überblick an. Hier sind eine Vielzahl von Enzymen beteiligt, die wir später noch genauer thematisieren.
Bevor die Verdopplung der DNA starten kann, muss die Doppelhelix zunächst entspiralisiert und die Wasserstoffbrückenbindungen zwischen den jeweiligen Basenpaaren getrennt werden. Es paaren sich hier immer nur die DNA Basen Adenin mit Thymin und Guanin mit Cytosin (=komplementäre Basenpaarung; Watson Crick Basenpaarung).
Nach der Öffnung der DNA liegen die beide Einzelstränge (Matrizen) voneinander getrennt vor. Jetzt können sich neue Nukleotide(=Zucker + Base + Phosphatgruppe), die im Cytoplasma hergestellt werden, an die Einzelstränge anlagern. Hier gilt natürlich auch das Prinzip der komplementären Basenpaarung.
Wie du bereits gelernt hast, erhalten wir nun 2 identische DNA Doppelstränge, die aus einem „alten“ und einem „neuen“ Einzelstrang zusammengesetzt sind.
Antiparallele DNA Stränge
Wichtig für das Verständnis der Replikation der DNA ist, dass wir uns eine Eigenschaft des DNA Aufbaus noch einmal vor Augen führen: die antiparallele Verlaufsrichtung der jeweiligen Einzelstränge zueinander. Sie sind also entgegengesetzt angeordnet, wodurch sich auch die Leserichtung der beiden Einzelstränge unterscheidet.
Ein Strang verläuft vom 5′-Ende (sprich: 5 Strich) zum 3′-Ende, der andere vom 3′-Ende zum 5′-Ende. Unter dem 5′-Ende kannst du das 5. Kohlenstoffatom des Zuckers mit seiner freien Phosphatgruppe und unter dem 3′-Ende das 3. Kohlenstoffatom mit der freien Hydroxylgruppe (OH-) verstehen.
Neue Nukleotide können immer nur an einem 3′-Ende angefügt werden, da nur an dieser Stelle eine freie OH-Gruppe vorliegt, die eine Bindung mit der Phosphatgruppe eines neuen Nukleotids eingehen kann. Das musst du bei der DNA Verdopplung unbedingt im Hinterkopf behalten!
DNA Replikation Ablauf im Detail
Schauen wir uns nun den molekularen Mechanismus der DNA Replikation Schritt für Schritt an. Du kannst die DNA Replikation in 3 Phasen unterteilen:
- Initiation
- Elongation
- Termination
Anschließend findet eine DNA Korrektur statt, die eventuell entstandene Fehler der DNA Kopien behebt. Das soll die Bildung von Mutationen verhindern.
Der allgemeine Ablauf der Replikation ist bei Prokaryoten (Bakterien und Archaeen ) und Eukaryoten sehr ähnlich. Allerdings kommen auch einige Unterschiede aufgrund der Größe und Komplexität der jeweiligen Genome (=Gesamtheit der DNA) vor. Auf diese gehen wir aber in einem späteren Abschnitt genauer ein.
Hier erklären wir dir nun den Ablauf der einzelnen Replikationsphasen im Detail:
Initiation
In der ersten Phase – der Initiation (lat. initare = beginnen) – startet die Replikation. Sie beginnt an definierten Stellen, die du als Replikationsursprung (origin of replication) bezeichnen kannst. Unter einem Replikon (Replicon) kannst du einen DNA-Abschnitt verstehen, der einen einzelnen Replikationsursprung enthält.
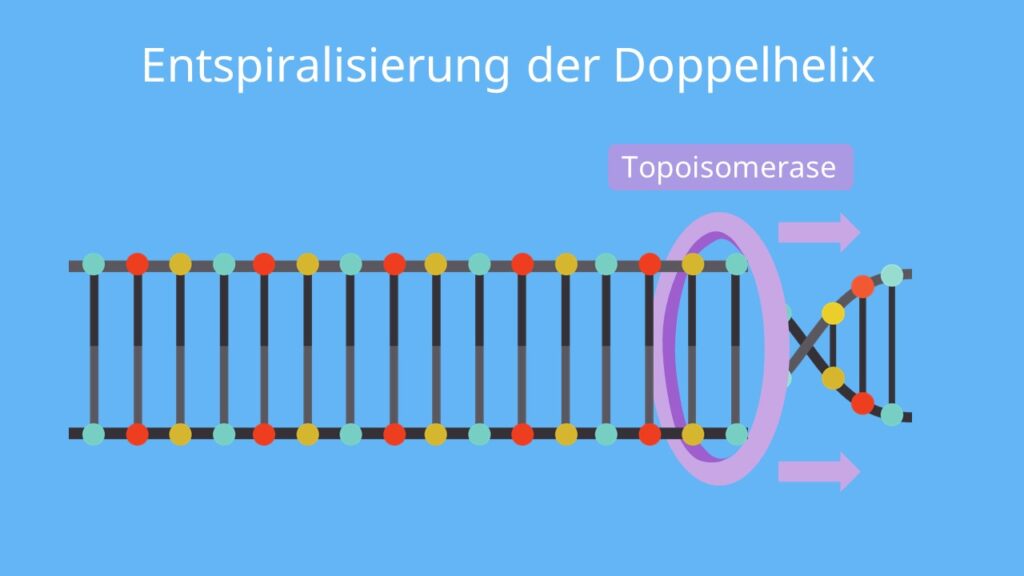
Schritt 1: Entspiralisierung der Doppelhelix
Zunächst bewirkt ein Enzym namens Topoisomerase, dass die DNA Doppelhelix entspiralisiert wird. Darunter kannst du verstehen, dass die Schraubenform quasi in eine Strickleiterform umgewandelt wird. Wichtig: Wir haben es hier aber immer noch mit einem Doppelstrang zu tun!
Schritt 2: Öffnung des DNA-Doppelstrangs
Um nun die beiden Einzelstränge zu erhalten, die als Vorlage (Matrize) für die neuen DNA Stränge dienen sollen, müssen zunächst die Wasserstoffbrückenbindungen zwischen den einzelnen Basenpaaren getrennt werden. Das kannst du auch als Denaturierung bezeichnen. Hierfür ist das Enzym DNA Helikase zuständig, das den Doppelstrang unter Energieverbrauch (ATP ) wie eine Art Reißverschluss öffnet.
Die Y-förmige Stelle, an der die Helikase die Wasserstoffbrückenbindungen löst, kannst du auch als Replikationsgabel bezeichnen. Es wandert von jedem Replikationsursprung aus je eine Replikationsgabel nach rechts und eine nach links (=bidirektionale Replikation).
Um die getrennten Stränge zu stabilisieren und zu verhindern, dass sich die beiden Stränge wieder aneinanderlagern, lagern sich spezielle Proteine (einzelstrangbindende Proteine, SSB-Proteine) an die jeweiligen Abschnitte.
Schritt 3: Anlagerung eines RNA-Primers
Damit die Replikation beginnen kann, sind sogenannte Primer („Startmoleküle“) notwendig, die von dem Enzym Primase hergestellt werden. Es handelt sich hier um ein kurzes RNA -Stück, das aus wenigen Nukleotiden besteht. Sie werden jeweils am 3′ Ende der Matrizenstränge befestigt.
Merke: Die kurzen RNA (Ribonukleinsäure)-Stücke (Primer) sind anders aufgebaut als die DNA: Sie enthalten statt der Base Thymin nämlich die Base Uracil und haben einen anderen Zucker in ihrem Gerüst. Daher müssen diese Stücke im späteren Verlauf der DNA Replikation ausgetauscht werden.
Elongation
Nach der Initiation kann nun die Elongation, also die Synthese neuer jeweils passender Einzelstränge (Tochterstränge), starten. Das Enzym, das für die Anheftung der einzelnen komplementären DNA-Basen zuständig ist, ist die DNA Polymerase.
Sie fügt neue Nukleotide an das 3′ Ende des Primers, denn nur dort ist eine Kettenverlängerung (Polymerisation) möglich. Die DNA Polymerase arbeitet also nur von 5′ → 3′ Richtung in Bezug auf die neu herzustellenden DNA Stränge.
Leitstrang Folgestrang
Ein Strang ist so orientiert, dass sein 3′ Ende ohne Unterbrechung verlängert werden kann, da die DNA Polymerase in die gleiche Richtung wie die Helikase arbeitet. Du kannst diesen Strang auch als Leitstrang bezeichnen. Es findet hier also eine kontinuierliche Verlängerung statt.
Der andere entstehende Strang — der Folgestrang — hingegen ist so angeordnet, dass sich sein zugängliches 3′ Ende von der Replikationsgabel entfernt und eine immer größer werdende Lücke entstehen würde. DNA Polymerase und Helikase arbeiten hier also in eine entgegengesetzte Richtung.
Doch es gibt eine Lösung: Die Primase (RNA Polymerase ) fügt immer weiter Primer an den Folgestrang an. Dadurch kann die DNA Polymerase also immer wieder von 5′ zu 3′ Richtung arbeiten (5′ → 3′). Sie hängt so lange neue Nukleotide an, bis sie den Primer des vorherigen Abschnitts erreicht hat. Hier erfolgt also eine diskontinuierliche (abschnittweise) Verlängerung mit Lücken.
Die dabei entstehenden kurzen DNA-Abschnitte kannst du auch als Okazaki Fragmente bezeichnen.
Nun kann auch der Austausch der RNA-Nukleotide des Primers erfolgen: Sie werden zunächst durch die RNase H entfernt. Daraufhin ersetzt eine weitere DNA-Polymerase die RNA-Nukleotide durch komplementäre DNA-Nukleotide.
Im letzten Schritt schließt das Enzym DNA Ligase die zwischen den jeweiligen Okazaki Fragmenten entstandenen Lücken wie eine Art Kleber.
DNA Replikation Enzyme
Hier haben wir für dich die wichtigsten an der DNA Replikation beteiligten Enzyme und ihre Funktionen in einer Tabelle zusammengestellt:
| Enzym | Funktion |
| Topoisomerase | Entwindung der Doppelhelix |
| Helikase | Öffnung der Doppelstränge durch Trennung der Wasserstoffbrückenbindungen zwischen den komplementären DNA Basen |
| Primase | Synthese eines RNA-Abschnitts zum Start der Replikation (Primer) |
| DNA Polymerase | DNA-Synthese am 3′ Ende durch das Anfügen komplementärer Nukleotide an die jeweiligen Einzelstränge |
| RNase H | Entfernung des RNA-Primers aus der neu hergestellten DNA |
| DNA Ligase | „Kleberenzym“: Verknüpfung der gebildeten Stränge |
DNA Replikation Eukaryoten
Die Replikation der DNA von Eukaryoten und Prokaryoten ist sich im Mechanismus sehr ähnlich. Allerdings musst du berücksichtigen, dass die DNA bei Eukaryoten im Zellkern viel dichter mit Proteinen (u.a. Histonen) verpackt vorliegt. Für die DNA Replikation, aber auch zum Ablesen der Gene (Proteinbiosynthese ), muss diese Verpackung aufgelockert werden.
Wie du bereits gelernt hast, besitzen Eukaryoten zudem viel komplexere Genome als Prokaryoten, was mehr Replikationsursprünge erfordert. Dadurch kann an den linear vorkommenden Chromosomen an mehreren Stellen gleichzeitig gearbeitet werden. Die menschliche DNA besitzt beispielsweise zehntausende Replikationsursprünge, die aber wahrscheinlich nicht alle aktiviert sind.
Außerdem verläuft die DNA Replikation bei Eukaryoten langsamer als bei Prokaryoten, denn die DNA Polymerasen können nur ca. 50-100 Nukleotide pro Sekunde verknüpfen. (Zum Vergleich: Bei den Prokaryoten schaffen die DNA Polymerasen mehr als 1000 Nukleotide pro Sekunde.) Diese Unterschiede kommen unter anderen deshalb zu Stande, da die Eukaryoten mehrere DNA-bindende-Proteine besitzen.
Beteiligte Enzyme
Außerdem besitzen die an der Replikation beteiligten Enzyme bei Eukaryoten und Prokaryoten meist die gleiche Funktionalität, aber einen unterschiedlichen Aufbau. So werden die eukaryotischen DNA Polymerasen nach griechischen Buchstaben (α, β, ..) benannt. Die Polymerase alpha (α), delta (δ) und epsilon (ε) sind für die Kettenverlängerung bei den eukaryotischen Lebewesen zuständig.
Auch die Primase kommt bei Eukaryoten nicht als eigenes Enzym, sondern an die Polymerase α gebunden vor.
Telomere zum Schutz der Chromosomenenden
Da die Genome der Eukaryoten aus linearen DNA-Molekülen bestehen, ergibt sich an den Enden des Folgestrangs ein Problem. Er kann nämlich nicht bis zum Ende synthetisiert werden, wenn der endständige Primer entfernt wird. Dadurch steht der DNA-Polymerase auch kein freies 3′-Ende zur Kettenverlängerung zur Verfügung.
Als Schutz vor Verlust von Erbmaterial besitzen eukaryontische Chromosomen an ihren Enden Sequenzwiederholungen – die Telomere. Sie selbst codieren für keine Proteine und werden durch das Enzym Telomerase hergestellt.
DNA Replikation Prokaryoten
Wie du bereits gelernt hast, besteht der größte Unterschied zwischen den Genomen von Eukaryoten und Prokaryoten darin, dass sie bei Prokaryoten kleiner und in zirkulärer Form vorkommen. Deswegen lässt sich hier auch meist nur ein Replikationsursprung finden. Bei dem Darmbakterium E. Coli handelt es sich beispielsweise um eine 250 Basenpaare lange Sequenz, die vor allem aus Adenin und Thymin besteht.
Die Replikation bei Prokaryoten verläuft, wie bereits erwähnt, viel schneller als bei den Eukaryoten.
Prokaryoten besitzen einen definierten DNA Abschnitt, der das Ende (Termination) der DNA Verdopplung einläutet. Er liegt gegenüber des Startpunkts. Am Ende der Replikation entstehen zwei identische ringförmigen Stränge. Sie liegen meist noch eine Weile an der terminalen Stelle ineinander verschränkt vor, bis das Enzym Topoisomerase die beiden DNA-Ringe voneinander trennt.
Die DNA Polymerasen in Prokaryoten werden nach römischen Zahlen (I – V) klassifiziert. Die Polymerase III für DNA Synthese zuständig.
Zusammenfassung
- Die DNA Replikation ist die identische Verdopplung des Erbguts und erfolgt immer vor der Kernteilung (Mitose )
- Es handelt sich (fast immer) um einen semikonservativen Mechanismus (jeder DNA Einzelstrang fungiert als Vorlage für die Synthese eines neuen Strangs)
- Die DNA Synthese kann in 3 Phasen (Initiation, Elongation und Termination) eingeteilt werden und startet immer am Replikationsursprung
- Das Enzym DNA Polymerase verknüpft neue Nukleotide in 5′ → 3′-Richtung; dabei entsteht ein Folge- und ein Leitstrang
Für mehr Informationen über die DNA Polymerase und ihre Rolle in der Biotechnologie, schaue dir gerne unsere Beiträge DNA Polymerase und Polymerase Kettenreaktion (PCR) an.