Restriktionsenzyme
Restriktionsenzyme sind Enzyme, die die DNA zerschneiden. Hier lernst du wie genau sie funktionieren und wie sie in der Gentechnik eingesetzt werden können! Du willst das Thema noch schneller erklärt bekommen? Dann schau dir hier das Video dazu an!
Inhaltsübersicht
Restriktionsenzyme einfach erklärt
Unter einem Restriktionsenzym verstehst du ein Enzym, das bestimmte DNA Sequenzen erkennen und schneiden kann. Du kannst es dir daher auch als molekulare Schere vorstellen, welche die DNA ganz gezielt zerschneidet. Ein anderer Name ist Restriktionsendonuklease, weil die Enzyme Bindungen in der DNA (= Nukleinsäure) spalten. Restriktionsenzyme dienen in Bakterien als ein natürliches Abwehrsystem gegen angreifende Viren. Der künstliche Einsatz in der Klonierung im Bereich der Gentechnik spielt aber auch eine große Rolle.
Restriktionsenzyme (auch Restriktionsendonukleasen, engl. restriction enzyme) sind Enzyme, die DNA Sequenzen sehr spezifisch erkennen und schneiden. Sie sind wichtige Werkzeuge in der Molekularbiologie.
Restriktionsenzyme Bedeutung
Die Restriktionsenzyme sind von Bedeutung, wenn es darum geht DNA zu schneiden. Das heißt, dass sie den DNA-Doppelstrang genau zwischen zwei Basenpaaren auseinander schneiden können. Sie zeichnen sich dadurch aus, dass sie sehr spezifisch arbeiten. Die Schnittstelle wird dabei anhand einer Erkennungssequenz bestimmt. Die Nukleasen erkennen Sequenzen, die 4-8 Basenpaare lang sind und oft eine sogenannte palindromische Sequenz aufweisen. Ein Palindrom ist ein Wort, welches vorwärts und rückwärts gelesen gleich ist, wie zum Beispiel Otto. In Bezug auf den DNA-Doppelstrang sind beide Stränge aus der selben Richtung (z.B. 5′-Richtung) gelesen gleich. Wie genau das aussieht, kannst du hier sehen:

|
5′-GAATTC-3′ | |
| 3′-CTTAAG-5′ |
 |
Die Genauigkeit der Enzyme macht man sich in der Gentechnik zu Nutze. So kann man zum Beispiel bei der Klonierung gezielt DNA Sequenzen zerschneiden und neu zusammenbauen. Bei Bakterien (Prokaroyten ) dienen die Restriktionsenzyme zur Abwehr gegen Viren (Bakteriophagen). Das kannst du dir ähnlich wie das Immunsystem von uns Menschen vorstellen. Fremde DNA wird von den Nukleasen direkt erkannt und abgebaut. Dabei ist es wichtig, dass die Bakterien zwischen eigener und fremder DNA unterscheiden können. Das machen sie über die Methylierung ihrer eigenen DNA. Das kannst du dir wie eine Art Markierung am DNA-Strang vorstellen.
Studyflix vernetzt: Hier ein Video aus einem anderen Bereich
Restriktionsenzyme Klassifizierung
Du kannst die Restriktionsenzyme anhand ihrer Eigenschaften in vier verschiedene Gruppen einteilen. Die Aufteilung in die Typen I-IV erfolgt je nach Aufbau und Funktionsweise der jeweiligen Enzyme. Beispielsweise die Festlegung der Schnittstelle anhand der Erkennungssequenz ist unterschiedlich. Eines der bekanntesten Typ II Enzyme ist EcoRI aus dem Bakterium E. coli.
Typ I
Typ I Restriktionsenzyme schneiden die DNA zufällig, weit weg von ihren Erkennungssequenzen. Du kannst sie dir als komplexe Enzyme mit mehreren Untereinheiten vorstellen. Um zu funktionieren, benötigen sie Energie in Form von ATP . Neben der Restriktion können sie auch Modifikationen übernehmen. Das bedeutet in dem Fall, dass sie Methylgruppen übertragen können.
Typ II
Typ II Endonukleasen schneiden die DNA nahe oder innerhalb ihrer Erkennungssequenz. Daher erhältst du hier genau definierte Schnittprodukte. Zerschnitten werden dabei palindromische Sequenzen, die bis zu acht Basenpaare lang sind. Dafür benötigt Typ II keine Energiezufuhr. Es ist die Klasse, die am häufigsten vorkommt und die einzige die momentan im Labor eingesetzt wird. Ihre Einsatzgebiete liegen vor allem in der Gentechnik (Klonierung) oder in der epigenetischen Forschung, zur Erkennung spezieller Krankheiten.
Typ III
Typ III Restriktionsenzyme sind wie Typ I Kombinationsenzyme mit Restriktions- und Modifikationsfunktion. Sie schneiden die DNA ca. 20-25 Basenpaare von der Erkennungssequenz entfernt und benötigen dazu ebenfalls ATP. Die Erkennungssequenz muss dabei zweimal vorkommen und entgegengesetzt orientiert sein.
Typ IV
Die Restriktionsenzyme von Typ IV schneiden nur modifizierte (z.B. methylierte) DNA. Bisher hat man Enzyme dieser Gruppe hauptsächlich im Bakterium E. coli gefunden.
Beispiel EcoRI
Einer der bekanntesten Typ II Restriktionsenzyme ist EcoRI. Der Name klingt für dich vielleicht etwas ungewöhnlich, lässt sich aber einfach erklären. Die Namen geben nämlich die Herkunft der Enzyme an. Die ersten drei Buchstaben stehen für die Art: Eco steht hier für das Bakterium Escherichia coli. Dahinter folgt ein Buchstabe für den Stamm, also R. Die römische Zahl eins am Ende sagt dir, dass es das erste Enzym ist, das in E. coli gefunden wurde.
Restriktionsenzyme in der Gentechnik
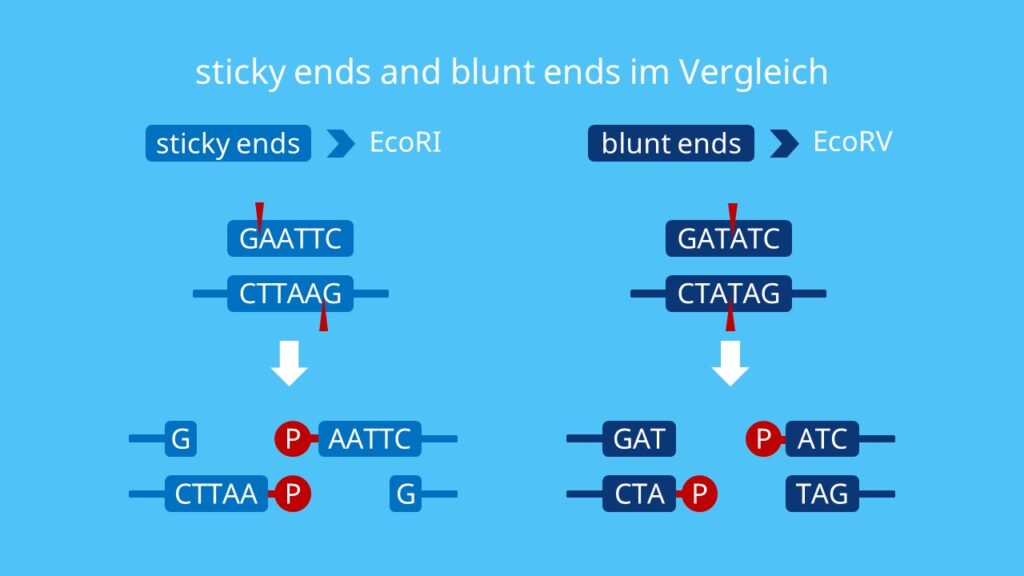
Als molekulare Werkzeuge gibt es für Restriktionsenzyme ein großes Einsatzgebiet in der Gentechnik. Am häufigsten setzt man sie für die DNA-Klonierung ein (Typ II). Darunter kannst du dir die identische Vervielfältigung von DNA-Sequenzen vorstellen. Dazu baut man DNA-Stücke zunächst in ein Plasmid ein. Ein Plasmid ist ein ringförmiges DNA-Molekül, dass dann als Vektor für den Transport, beispielsweise in Bakterien dient. Damit der Einbau funktioniert ist es wichtig, dass man Plasmid und einzubauende DNA (= Insert) mit dem gleichen Restriktionsenzym schneidet. Das nennst du Restriktionsverdau. Dazu mischst du die zu schneidende DNA Sequenz mit dem Restriktionsenzym in einer entsprechenden Pufferlösung . So wird die DNA an der Erkennungssequenz geschnitten. Das verwendete DNA-Stück kann dabei beispielsweise direkt aus einem Genom stammen oder das Produkt einer PCR sein. Je nach Schnittweise des Enzyms entstehen entweder sticky ends (klebrige Enden) oder blunt ends (stumpfe Enden). Bei sticky ends wird versetzt geschnitten, so dass überlappende Enden mit komplementären Basen entstehen. Blunt ends entstehen durch gerade Schnitte.
Im zweiten Schritt kannst du verschiedene Enden, die mit dem gleichen Enzym geschnitten wurden mit Ligasen wieder zusammenkleben (= Ligation). So erhältst du dann dein Plasmid mit dem zusätzlichen DNA Abschnitt (= Insert).
Mithilfe des Restriktionsverdaus lassen sich aber auch Restriktionsanalysen durchführen. Darunter verstehst du die Charakterisierung von DNA durch gezielte Spaltung. Die Fragmente die beim Verdau entstanden sind, werden durch eine Gelelektrophorese nach ihrer Größe aufgetrennt. So kannst du zum Beispiel Rückschlüsse über die Größe und den Aufbau von Plasmiden oder die Position von Schnittstellen ziehen. Anhand der Fragmentlänge ist es möglich Tierarten oder Viren genetisch voneinander zu unterscheiden.
Wurde dein Interesse geweckt und du möchtest noch mehr zum Thema Klonierung erfahren? Dann schau dir einfach dieses Video an!
Restriktionsenzyme — häufigste Fragen
(ausklappen)
Restriktionsenzyme — häufigste Fragen
(ausklappen)-
Was sind Restriktionsenzyme?Restriktionsenzyme, oder auch Restriktionsendonukleasen (REN), sind kleine Proteine. Sie erkennen spezielle DNA-Sequenzen und können gezielt an ihnen schneiden. Deshalb werden sie auch molekulares Schneidewerkzeug genannt. Die Ligasen sind die Antagonisten der Restriktionsenzyme.
-
Was ist ein Southern Blot?Der Southern Blot ist ein molekularbiologisches Verfahren, das spezifische DNA-Sequenzen in DNA-Proben nachweisen kann. Dafür spalten die Restriktionsenzyme die DNA in einzelne Teile.